背景
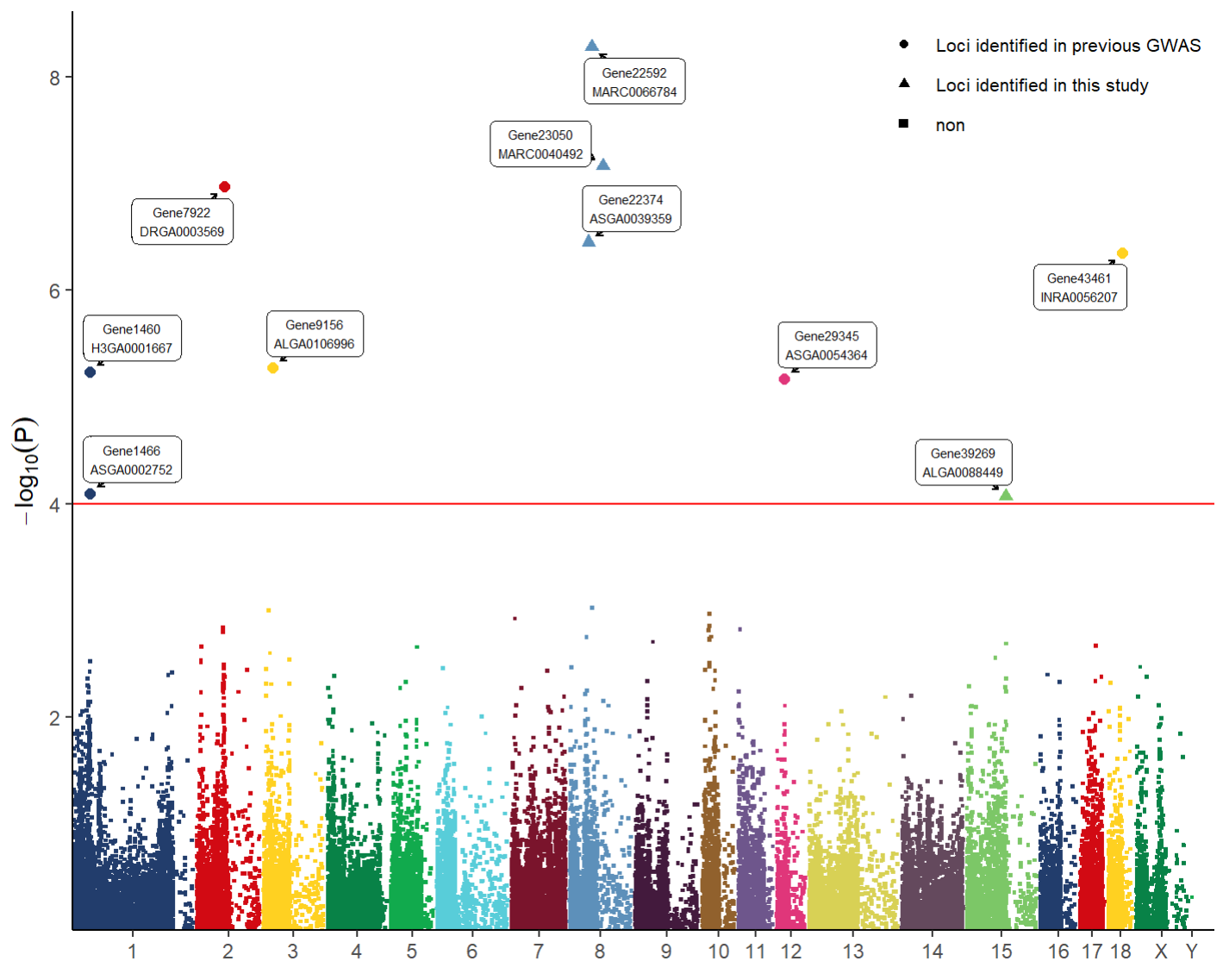
画出paper里的曼哈顿图,文字标签好看些。
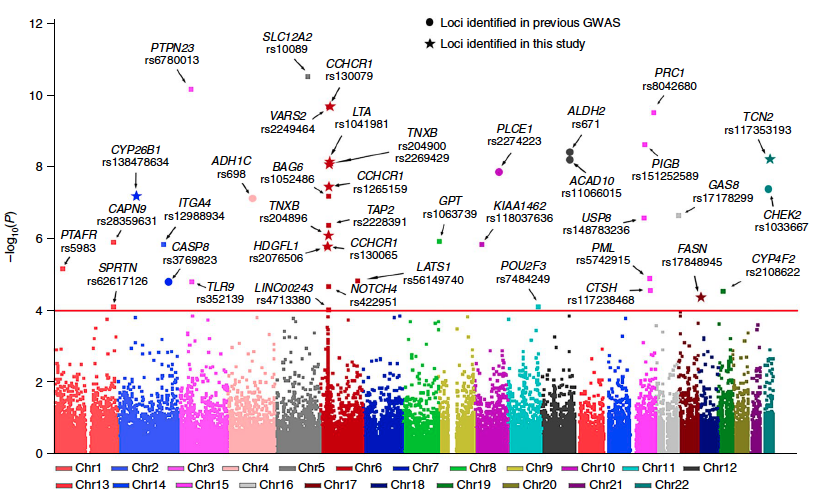
出自文章PMID: 29379198
应用场景
展示每条染色体上的SNP分布,通常作为外显子组测序文章里的第一个图。
输入文件
easy_input.csv,包括染色体名字、SNP编号、在染色体上的位置(第几个碱基)、pvalue、类型type、基因名gene。
其中type和gene,非必需,如果能提供基因名,就写在gene列;如果要用不同形状来展示不同类的SNP,会把类型写到type。
library(dplyr)
gwas <- read.csv("easy_input.csv", as.is = T)
head(gwas)
str(gwas)
gwas$chr <- factor(gwas$CHR,levels=c(1:18,"X","Y"))#给染色体排序
如果自己不提供染色体长度,就用SNP位置最大值作为染色体长度,运行下面这段
chrx <- gwas %>%
group_by(chr) %>%
summarise(chr_len=max(POS)) %>%
#计算每条染色体在x轴上的起始位置
mutate(chrx=cumsum(chr_len)-chr_len) %>%
select(-chr_len) %>% #删除染色体长度
data.frame()
Karyotype.txt,如果你自己提供染色体长度文件,就运行下面这段
chrom <- read.table("Karyotype.txt", header = T, as.is = T)
head(chrom)
#计算每条染色体在x轴上的起始位置
chrom$chrx <- c(0,cumsum(as.numeric(chrom$chr_len))[-nrow(chrom)])
chrx <- data.frame(chr = chrom$chr, chrx = chrom$chrx)
chrx$chr <- factor(chrx$chr,levels=c(1:18,"X","Y"))#给染色体排序
计算每个SNP的坐标
#计算每条染色体在x轴上的坐标
gwas <- left_join(gwas, chrx, by=c("chr"="chr")) %>%
#arrange(chr, POS) %>%
mutate(SNPx=POS+chrx)
#取-log10(P-value)作为y轴坐标
gwas$SNPy <- -log10(gwas$P_value)
用阈值分类
#设置阈值
logPcutoff <- 4
#是否超过阈值分为两类,后面将用不同大小的点来区分
gwas$pcut <- rep("0", nrow(gwas))
gwas[gwas$SNPy > logPcutoff,]$pcut <- "1"
#只给超过一定阈值的SNP写文字标签
gwas$label <- rep(NA, nrow(gwas))
gwas[gwas$SNPy > logPcutoff,]$label <- paste(gwas[gwas$SNPy > 4,]$gene,gwas[gwas$SNPy > 4,]$SNP,sep = "\n")
#如果没有提供基因名,就用下面这行
#gwas[gwas$SNPy > logPcutoff,]$label <- gwas[gwas$SNPy > 4,]$SNP
tail(gwas)
str(gwas)
开始画图
先画点
#不喜欢默认的配色,自定义足够多的颜色
mycol <- rep(c("#223D6C","#D20A13","#FFD121","#088247","#11AA4D","#58CDD9","#7A142C","#5D90BA","#431A3D","#91612D","#6E568C","#E0367A","#D8D155","#64495D","#7CC767"),2)
#染色体的名字作为x轴标签
axisdf = gwas %>% group_by(chr) %>% summarize(center=( max(SNPx) + min(SNPx) ) / 2 )
library("ggplot2")
p <- ggplot(data = gwas, mapping = aes(x = SNPx, y = SNPy)) +
geom_point(aes(colour = chr, #每条染色体用不同的颜色
shape = type, #不同类型用不同形状
size = pcut)) + #阈值上下的点用不同大小
#如果没有分类信息,或者不需要用不同的形状显示分类,就运行下面这行
#geom_point(aes(colour = CHR)) +
scale_size_discrete(range = c(0.5,2)) + #点的大小范围
#scale_shape_manual(values = c(20,8,15)) + #点的形状
#添加阈值的红色直线
geom_hline(yintercept = logPcutoff, color = 'red') +
xlab("") + ylab(expression(-log[10](P))) +
scale_x_continuous(label = axisdf$chr, breaks= axisdf$center,
expand = expand_scale(mult = c(0,0.02))) + #左边到头,右边留空
scale_y_continuous(expand = expand_scale(mult = c(0,0.04))) + #下面到头,上面留空
theme_bw() + #去除背景色
theme(panel.grid =element_blank()) + #去除网格线
theme(panel.border = element_blank()) + #去除外层边框
theme(axis.text = element_text(size = rel(0.8)), #坐标轴标签字号
axis.line = element_line(colour = "black")) #沿坐标轴显示直线
p1 <- p + scale_color_manual(values = mycol) + #使用自定义的颜色
guides(colour = FALSE, size = FALSE) + #不显示染色体图例
theme(legend.background = element_blank(),
legend.title = element_blank(),
legend.text = element_text(size = 8), #图例字号
legend.position = c(1,1),legend.justification = c(1,1)) #形状的图例放在右上角
p1
#图例放到底部
p2 <- p + theme(axis.ticks.x = element_blank(), axis.text.x = element_blank()) +#去除x轴刻度和标签
guides(color = guide_legend(nrow = 2), #染色体的图例排两行
size = FALSE) + #不显示点大小的图例
theme(legend.background = element_blank(),
legend.title = element_blank(),
legend.box = "vertical", #两个图例垂直摆放
legend.margin=margin(t= 0, unit='cm'), #图例不留边
legend.spacing = unit(0,"in"), #两个图例紧挨着
legend.position = "bottom") + #图例放在底部
scale_color_manual(labels = paste0("Chr",unique(gwas$chr)),
values = mycol)
p2
p1:
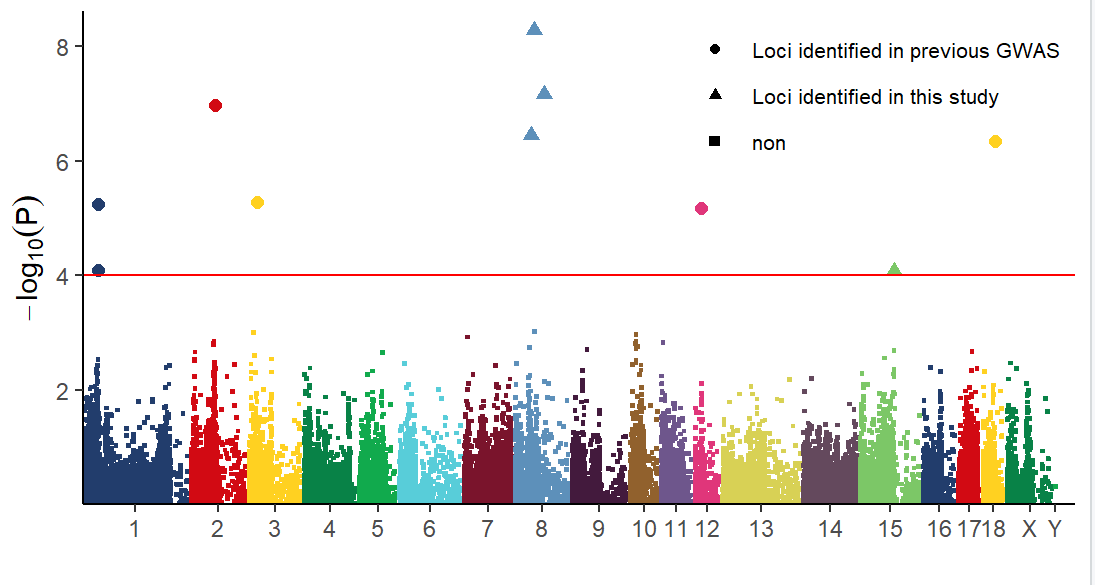
P2:
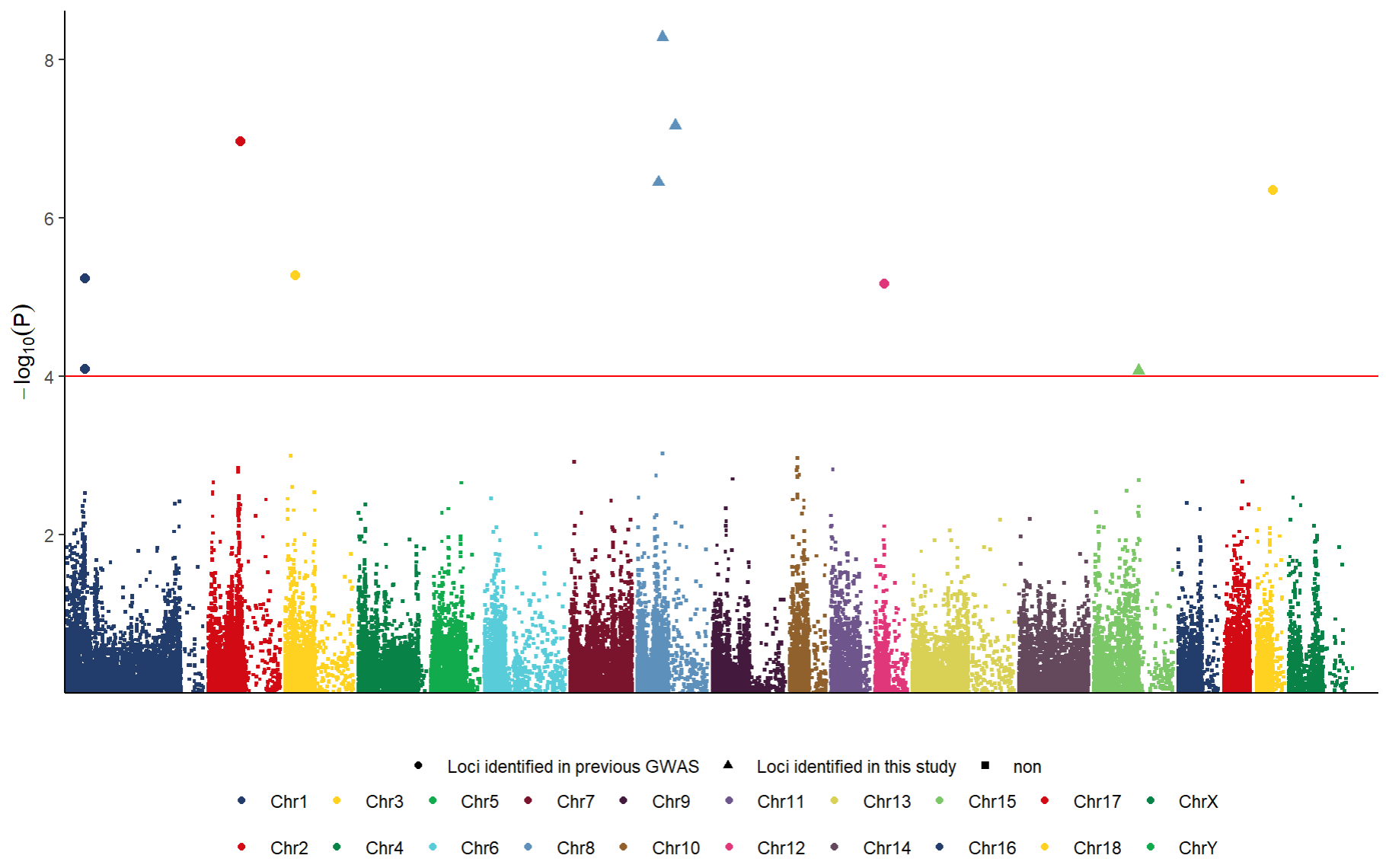
再加箭头和标签
library(ggrepel)
p2 + geom_text_repel(aes(label = label),
size = 2, #标签文字的字号
point.padding = unit(0.5, "lines"), # label与point之间的连接显示
segment.colour = "black", # 连接(直线+箭头)的颜色
segment.size = 0.4, # 连接(直线+箭头)的粗细
arrow = arrow(angle = 20, length = unit(0.04,"inches")), # 箭头设置参考?arrow
ylim = c(logPcutoff, max(gwas$SNPy)), # 限制label的绘图区域
direction = "both", # 默认在x、y轴方向上调整label的位置
min.segment.length = 0, #默认长度小于0.5的箭头就不画了
na.rm = TRUE #不提示移除NA
)
#保存到pdf文件
ggsave("Manhattan.pdf",width = 7,height = 4)
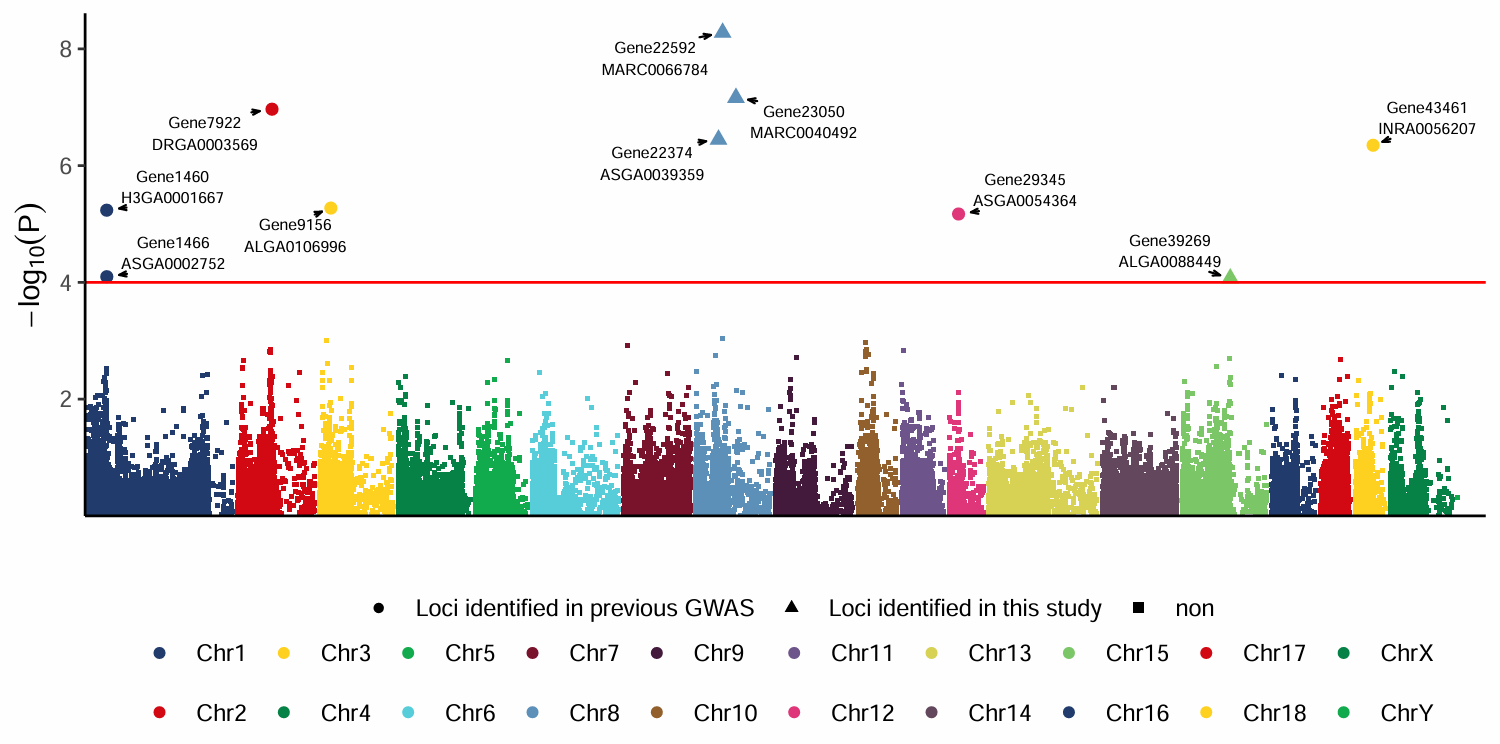
还可以给标签加上边框
p1 + geom_label_repel(aes(label = label),
size = 2,
point.padding = unit(0.5, "lines"),
label.size = 0.1, # label外的边框宽度
label.r = unit(0.2, "lines"), # label外的边框直角弧度
segment.colour = "black",
segment.size = 0.5,
arrow = arrow(length = unit(0.1,"cm")),
ylim = c(logPcutoff, max(gwas$SNPy)),
direction = "both",
min.segment.length = 0,
na.rm = TRUE
)
 |Archiver|手机版|小黑屋|bioinfoer
( 萌ICP备20244422号 )
|Archiver|手机版|小黑屋|bioinfoer
( 萌ICP备20244422号 )