1 数据来源
本次带来复现的图来自Nature Communications期刊的文章
《Host biology, ecology and the environment influence microbial biomass and diversity in 101 marine fish species》
正文Figure4a
)
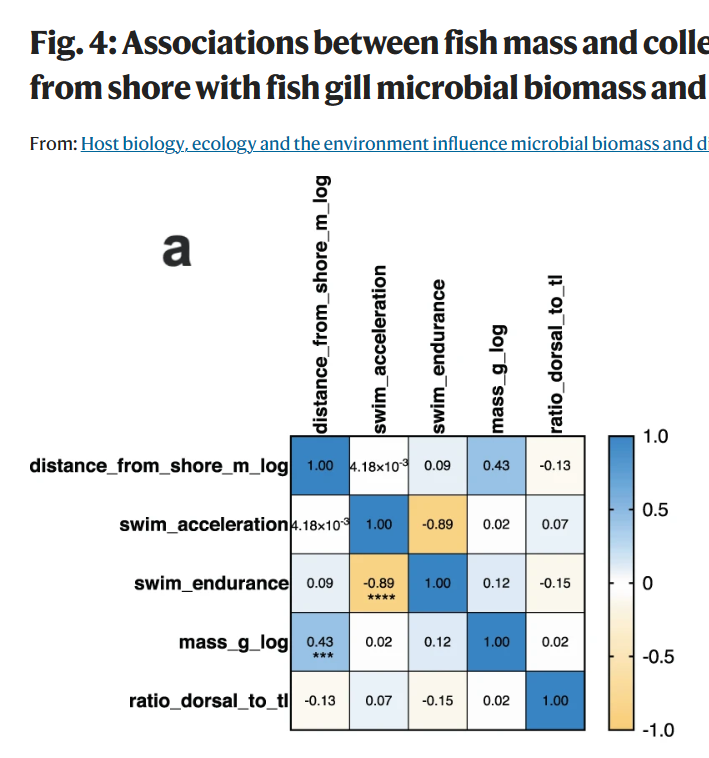
数据在作者的source data表格中。
当然后面代码和数据都会留给大家。
2 画图
直接上代码
rm(list = ls()) #### 魔幻操作,一键清空~
getwd()
setwd('C:/Users/Phil/公众')
library(ggplot2)
library(corrplot)
#数据输入
Corr_data = read.table('fig4a.txt', header=T, sep="\t", quote = "", row.names=NULL, comment.char="",stringsAsFactors = FALSE)
#相关性矩阵计算
Co_data = cor(Corr_data,use='pairwise.complete.obs',method='spearman')
p_values <- cor.mtest(Corr_data,conf.level=0.95,method='spearman')$p
# 打开一个PDF文件
pdf("corr_heatmap.pdf",width=8, height=6)
#绘图
#下三角矩阵
corrplot(Co_data,col = colorRampPalette(c("#FBD37D","white","#4784C1"))(100),tl.col = "black",
method = 'color',
type = 'lower',
is.corr = T,
cl.cex = 1,
tl.cex = 1.2,
cl.pos = 'n',
tl.pos = 'lt',
addCoef.col = 'black',
p.mat = p_values,
sig.level = c(.001,.005,.01,.05), #根据P值标出不显著,若用insig = "blank"将不显著的空白处理
insig = 'label_sig',
pch.cex = 1.5,
outline = 'black')
#上三角矩阵
corrplot(Co_data,col = colorRampPalette(c("#FBD37D","white","#4784C1"))(1000),tl.col = "black",
method = 'color',
add = T,
type = 'upper',
tl.pos = 'n',
is.corr = T,
cl.cex = 1,
cl.ratio = 0.25,
tl.cex = 1.2,
addCoef.col = 'black',
outline = 'black')
dev.off()
#保存的pdf使用ai打开调整重叠的星号等。
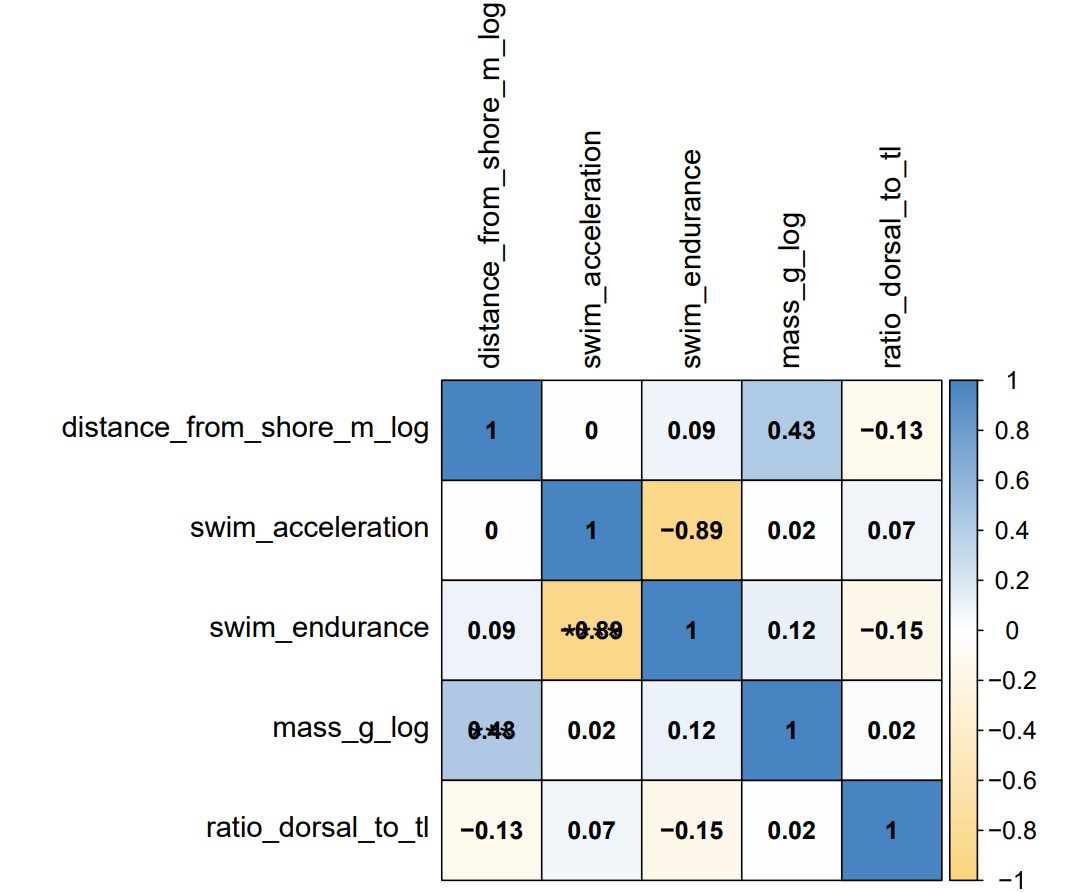
结果
)
代码中需要用到的输入数据:fig4a.txt以及全部代码,都可以在公众号"音云商城"后台回复“20240502。
 |Archiver|手机版|小黑屋|bioinfoer
( 萌ICP备20244422号 )
|Archiver|手机版|小黑屋|bioinfoer
( 萌ICP备20244422号 )