背景
最近用4.4.1的R有时候包装不上,可能rtools啥的编译有问题,试试另一种安装r的方式看看。
操作步骤
安装scoop
首先是在管理员运行Powershell里跑以下两行代码,安装scoop。
Set-ExecutionPolicy RemoteSigned -Scope CurrentUser
iex "& {$(irm get.scoop.sh)} -RunAsAdmin"
安装GNU核心工具
接下来,就可以用scoop来安装各种软件了,又是两行代码,这次安装的是GNU的核心工具,包括bash, make, gawk and grep等等。
scoop bucket add main
scoop install coreutils
安装R
一条指令搞定:
scoop install r
做为R开发者,你还需要点啥 (Optional)
这是可选项,因为R语言很多人只是用户。而做为开发者的话,我们还需要有rtools,和pandoc。latex
用以下指令安装就行:
scoop install rtools
scoop install pandoc
scoop install pandoc-crossref
安装pandoc是编译vignettes时需要。而你编译R包的话,是需要rtools的。
Rstudio配置
Tools-Global options中选择
C:\Users\Administrator\scoop\apps\r\4.5.0\bin\R.exe
然后重启就可以
配置rtools,发现是自动匹配的那就不用弄了。
Sys.which("make")
# "c:\\rtools45\\usr\\bin\\make.exe"
##查看r包安装位置
.libPaths()
# [1] "C:/Users/Administrator/scoop/persist/r/site-library"
# [2] "C:/Users/Administrator/scoop/apps/r/4.5.0/library"
[1] # 用户级包路径(scoop管理)
[2] # R系统自带包路径
一般不用修改。
建立R包下载目录
安装一开始会先下载包,这些临时文件默认是存储在C盘的某个路径下,C盘不大的话,我建议改动下这个地方。
具体方法就是先建立临时文件存储的路径例如 C:\Users\Administrator\software\R\RStudiowork\R-4.5.0\downloaded_packages
检验rtools是否成功
install.packages("jsonlite", type = "source")
原命令是这个样子,我们需要改动参数
我们执行
options("repos" = c(CRAN="https://mirrors.tuna.tsinghua.edu.cn/CRAN/"))
install.packages("jsonlite", type = "source",destdir = 'C:/Users/Administrator/software/R/RStudiowork/R-4.5.0/downloaded_packages')
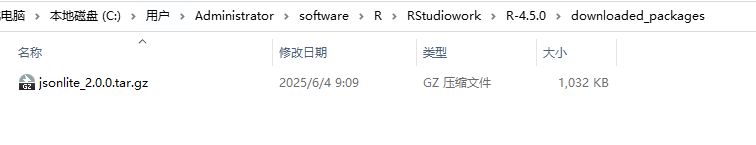
安装成功,下载目录也有记录
好了,本次教程到此结束。
后记
使用scoop管理r软件更简单了,不过此次仅试验,因为结合了rstudio去使用,不知道后续有无问题,正常可以直接使用vscode以及bash去使用,不过因为笔者是win10,bash不知道为啥用不起来(win11可以自定义配置文件,从而使用bash终端)。
检查对应的目录中也没有bash.exe.
所以就先这样吧,用起来,出问题再说。
 |Archiver|手机版|小黑屋|bioinfoer
( 萌ICP备20244422号 )
|Archiver|手机版|小黑屋|bioinfoer
( 萌ICP备20244422号 )